Experimenta
En el marco del proyecto “Saca La Lengua” se utiliza un vocabulario raro y específico, que incluye palabras que pueden resultar poco comunes. Esto es porque se trata de palabras que se suelen utilizar solamente en el contexto de un estudio científico.
¿Quieres saber qué significan? ¿De qué conceptos estamos hablando?
Verás que no es nada complicado, a veces te propondremos vídeos y juegos para intentar familiarizarte con estos conceptos nuevos.
¡Qué disfrutes de tu ronda de descubrimiento!
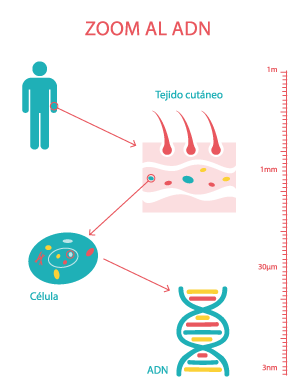
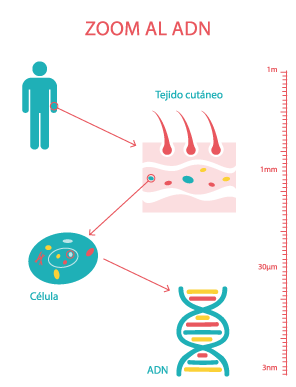
1 El ADN
El ADN (iniciales de ácido desoxirribonucleico) se encuentra en el núcleo de la célula.
El ADN está formado por un azúcar, un grupo fosfato y 4 bases nitrogenadas distintas:
• adenina → A,
• timina → T,
• citosina → C
• guanina → G
Todos estos elementos juntos forman una doble hélice o escalera que gira sobre si misma.
La forma de doble hélice del ADN fue descubierta por James Watson y Francis Crick en 1953 (ver su publicaciones originales aquí). Por este descubrimiento recibieron el Premio Nobel en 1962 conjuntamente con Maurice Wilkins. Sin embargo, se debate si Rosalind Franklin no debería haber recibido también este reconocimiento por el descubrimiento.

Utilizando tan solo 4 “letras distintas” se escribe “el libro” del ADN
Este “libro” contiene todas las instrucciones para construir todos y cada uno de los elementos necesarios que componen un ser vivo, como un humano, un gato o una planta: todos tenemos ADN formado por 4 “letras”.
Las partes de lectura del ADN se copian en el ARN y, generalmente, éste las traslada a cadenas de aminoácidos o proteínas (el principal material de construcción de las células y, por tanto, de los seres vivos).
El ADN humano mide más o menos 2 metros.
Debemos tener en cuenta que la célula tiene un tamaño de unos micrómetros y, por ello, el ADN se encuentra compactado, muy enrollado sobre si mismo, gracias a una estructura formada por histonas (proteínas).
¿Quieres saber más?
Aquí encontrarás más información contextualizada.
Propuesta de actividades didácticas y lúdicas para conocer mejor el ADN
El National Centre for School Biotechnology propone construir una cadena de ADN en 3 dimensiones con papel.
La Universidad de Utah propone construir un modelo de ADN empaquetado gracias a las histonas. Las instrucciones se pueden encontrar en este enlace. Y puedes descargar el pdf para hacerlo en este enlace.
Juego en línea para construir una cadena de ADN.
Construye una cadena de ADN en forma de doble hélice.
Averigua el número de palabras que se pueden formar con los 4 nucleótidos del ADN.
El juego del detective de ADN, para jugar solo o con los compañeros de clase.
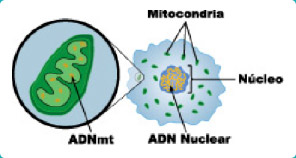
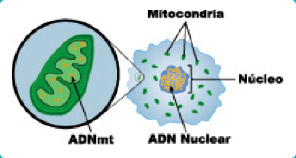
2 ADN no nuclear y ARN no codificante para proteínas
El ADN se encuentra en el núcleo de la célula. Pero hay ADN también en otros orgánulos (partes) de la célula.
Por ejemplo, podemos econtrar ADN en las mitocondrias. El ADN mitocondrial fue descubierto por Margit M. K. Nass y Sylvan Nass en 1963.
Advertirás que sólo hay 37 genes en el ADN mitocondrial humano frente a los 20.000 – 25.000 genes del ADN cromosómico nuclear de los humanos.
También se ha encontrado ADN en los cloroplastos de las plantas . Este ADN fue descubierto en 1962, y tiene más o menos 150 genes.
Todas las partes de lectura del ADN no sirven para hacer proteínas. Se ha comprobado que algunas partes del ADN que se copian a ARN no son traducidas posteriormente a proteínas. Este ARN, “no-codificante” juega un importante papel en el funcionamiento de la célula. Por ejemplo, participa en la construcción de la estructura de los ribosomas.
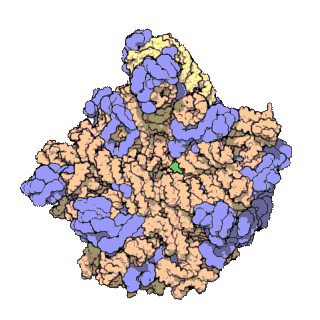
Los ribosomas son orgánulos que se encuentran dentro de la célula y están formados por proteínas (en azul) y ARN (en rosa, amarillo y verde).
El nombre de este ARN es el mismo que la región del ADN que codifica (que tiene la información) para hacer este ARN.
Por ejemplo, en las bacterias los ribosomas están hechos con ARN llamado 16S rDNA que viene de un trocito de ADN llamado “16S rDNA”.
O en los hongos, los ribosomas están hechos con ARN llamado ITS que vienen de un trocito de ADN llamado “ITS”.
¿Quieres saber más? ¡Prueba a hacerlo tú mismo!
3 Barcoding, para identificar especies
El término “barcoding” designa un procedimiento que consiste en escoger un fragmento de ADN estándar que sirva como sistema de identificación de especies y que, a la vez, facilite el descubrimiento de otras nuevas.
El término “barcoding” fue utilizado por primera vez por Paul Hebert de la Universidad de Gelph en 2003.

Esta técnica se basa en el estudio de un fragmento de ADN de más o menos 600 nucleótidos, de una misma especie. Tras la secuenciación de este trozo de ADN, se compara con fragmentos de ADN de la misma región de ADN que anteriormente habíamos secuenciado. Si se trata de la misma secuencia significará que estamos ante la misma especie. Si, por el contrario, la secuencia es diferente se puede suponer que es una especie nueva, por lo que será necesario proceder a realizar pruebas y análisis de datos de tipo filogenético.
En los animales, la referencia utilizada para realizar el “barcoding” es una parte del ADN mitocondrial que se llama el COI (el gen citocromo C oxidasa I).
Sin embargo, en el caso de las plantas, la referencia utilizada por el “barcoding” es una parte del ADN del cloroplasto que se llama rbcL y/o matK.
En las bacterias y los hongos, la referencia utilizada por el “barcoding” es un fragmento de ADN que codifica a ARN ribosomal. Se denomina:
• 16S rDNA para las bacterias
• Y ITS para los hongos
¿Quieres aprender más? ¡Hazlo tú mismo consultando la información a continuación!
El consorcio del “barcoding” para todas las especies vivas tiene un sitio web: ibol.org.
Este otro sitio web está dedicado especialmente al estudio de los hongos: fungalbarcoding.org
La Universidad de Cold Spring Harbor propone a sus estudiantes aplicar técnicas de “barcoding” para desarrollar sus propios proyectos de investigación.
4 Recogida de muestras
La recogida de muestras del proyecto Saca La Lengua en imágenes, ¡mira nuestra infografía!
El proyecto “Saca La Lengua” centra su interés en el estudio de los microorganismos que se encuentran en la boca.
Para poner en marcha este proyecto hemos probado 3 maneras o protocolos diferentes de recogida del ADN de los microorganismos.
1. Tubo para recoger saliva
Se escupe en un tubo, que se cierra y se envía al laboratorio para su análisis.
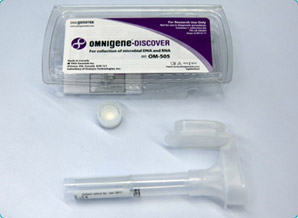
2. Algodón
Se frota el algodón contra la pared interior de la boca y se envía posteriormente al laboratorio donde se analizará.

Estos dos métodos son los utilizados mayoritariamente en estudios de ADN humano, animal o ADN mitocondrial.
Los resultados obtenidos, aplicando estos dos protocolos, han dado resultados para la amplificación del ADN de las bacterias, pero no en el caso del ADN de los hongos. Por lo tanto, se ha procedido a probar una tercera metodología para la recogida de saliva, descrita en el punto siguiente.
3. Enjuague bucal con agua salina
Centrifugadora

Teniendo en cuenta la literatura científica, este es el único método de recogida que, hasta ahora, ha dado resultados en el estudio de los hongos de la boca.
Este protocolo de extracción será utilizado en el proyecto “Saca la Lengua” e implica que, tras la recolección del material biológico bucal, es necesario proceder a centrifugar la muestra que se halla en el tubo, de forma que el material se concentre. A continuación, las muestras se trasladarán al laboratorio.
Un científico visitará los 40 puntos de recolección de muestras de España, adonde llevará la centrifugadora en una furgoneta para poder poner en práctica este protocolo.
5 Extracción de ADN
Friederich Mischer, biólogo y médico suizo (Basilea, 1844-1895), fue el primero en aislar e identificar ácidos nucleicos (ADN) de los leucocitos.
Los criterios que debe cumplir un método de extracción de ADN son:
– Eficiencia
– Suficiente ADN
– Eliminación de contaminantes
– Pureza y calidad del ADN
El método de extracción sigue estos pasos:

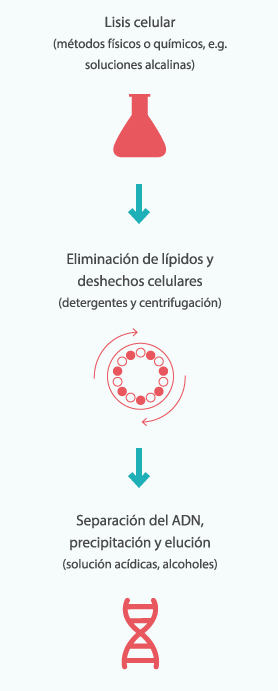
- Lisis celular: método que permite romper la membrana de la célula y la membrana nuclear (o mitocondria, o cloroplasto) para la liberación del ADN.
- Eliminación de lípidos y deshechos celulares con detergentes y centrifugación: el ADN se encuentra rodeado por proteínas (lípidos) que debemos quitar (limpiar) para tener más fácil “acceso” al ADN.
- Separación del ADN , precipitación y elución con soluciones ácidas o alcoholes para separarlo del medio acuoso, en donde se encuentran otros elementos pequeños. Así, obtendremos solamente el ADN.
¿Quieres aprender más? ¡Hazlo tú mismo consultando la información a continuación!
Tras una breve presentación, paso por paso, del protocolo de extracción de ADN, la Universidad de Utah propone que hagas el protocolo tú solo de forma virtual.
La Universidad de Cold Spring Harbor invita a realizar el protocolo (parte “procedure”) y ofrece más información sobre el mismo.
6 Amplificación del gen de interés – PCR
El método de reacción en cadena de la polimerasa (PCR, en sus siglas en inglés) es una técnica que permite obtener un gran número de copias de un fragmento de ADN. Esta técnica fue desarrollada por Kary Mullis, bioquímico estadounidense en 1987. Gracias a la técnica de PCR, Kary Mullis recibió el Premio Nobel de química en 1993.
La técnica de PCR ha revolucionado la investigación en biología molecular.
Para realizar numerosas copias de una parte del ADN es necesario:
- Una máquina capaz de cambiar la temperatura de las muestras rápidamente (90°C, 55°C, 72°C, 4°C): el termociclador.
-
Reactivos:
- ADN molde (que contenga la secuencia que nos interesa)
- Nucleótidos ( A, C, G, T)
- Iniciadores o cebadores o partidores (“primers” en inglés)
- ADN polimerasa
- Solución iónica y tampón (“buffer”)


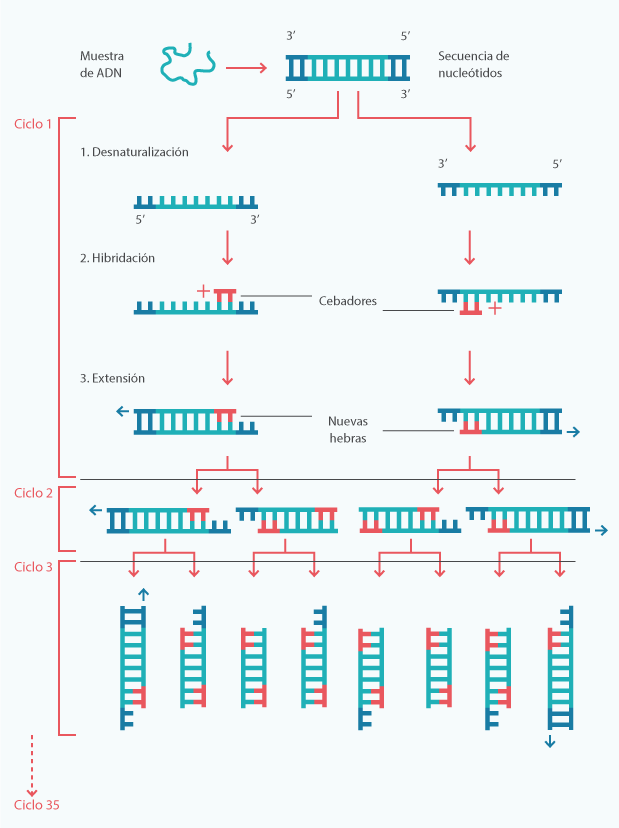
Los pasos principales de la PCR son
– 92°C : desnaturalización: las dos cadenas de ADN se separan
– 55°C: hibridación de los cebadores a un sitio muy específico de la cadena de ADN
– 72°C: extensión: la ADN polimerasa sintetiza nuevas cadenas de ADN complementarias
Así se obtiene el producto de PCR (amplificación de la secuencia de ADN que nos interesa).
7 Control de la amplificación del ADN o del producto de la PCR
a. Se utiliza la técnica de electroforesis
La electroforesis consiste en la separación de moléculas según su movilidad en un campo eléctrico. La electroforesis del ADN sirve para separar moléculas de ácidos nucleicos:
– En un medio viscoso y poroso (gel)
– Bajo un campo eléctrico
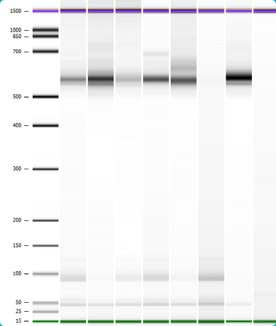
b. Se utiliza la técnica del “LabChip”
Este equipo de la compañía Caliper Life Science hace lo mismo que un gel de electroforesis pero de forma más eficiente y totalmente automatizada.
Si hubiéramos de analizar los resultados de las 2000 PCR de hongos mediante un gel de electroforesis tradicional, un técnico tardaría alrededor de 50 minutos por cada 12 muestras, que harían un total de 8333 minutos (139 horas) que equivaldrían a 18 jornadas de trabajo sin parar.
Gracias a la tecnología de microfluidos “Labchip”, ¡las 2000 electroforesis se hacen en tan solo 21 horas! ¡El técnico solo pone la placa con las muestras (96 cada vez) y la máquina lo hace todo sola!

- En primer lugar, se coloca un microlitro de la muestra que se analizará en cada uno de los 96 pocillos de la placa (¡este paso también se puede automatizar con una máquina!)
- Al colocar esta placa en la máquina con un chip especial, cada muestra viajará por separado dentro de microtubos cargados, lo que permite que las muestras se separaren por tamaño.
Este tecnología permite pasar de una electroforesis en formato de gel a un formato de “chips” con micro fluidos, lo que reduce el tiempo de obtención de resultados y la cantidad de reactivos necesarios para hacerlo.
Si quieres ver cómo funciona esta técnica, tienes este vídeo de la empresa Caliper Life Science.
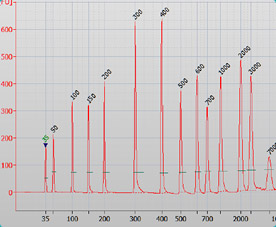
Los resultados se pueden leer en una imagen parecida a un gel de electroforesis o en un “electroferograma”.
¿Quieres conocer más? ¡Pruébalo tú mismo!
“El Centro de Regulación Genómica (CRG) ofrece la posibilidad de hacer una práctica con electroforesis en el aula. Los profesores interesados pueden solicitar el material necesario, que se le enviará de forma gratuita al centro. En este enlace se puede comprobar el calendario de disponibilidad de los kits y rellenar el formulario de petición.
Esta práctica permitirá hacer en el aula una simulación del trabajo de nuestros investigadores, y concretamente de la fase de comprobación de que la reacción de amplificación ha funcionado y que tenemos muchas copias del ADN de la región que queremos secuenciar.”
“La Fundación Pública Andaluza para la Investigación Biosanitaria en Andalucía Oriental-Alejandro” explica, paso por paso, la técnica de electroforesis en esta animación. Tras esta presentación, la Universidad de Utah propone que realices el protocolo de forma virtual.
8 Librería de ADN
Al secuenciar las muestras mediante técnicas de secuenciación de alto rendimiento, es necesario realizar un paso obligatorio que se denomina “librería de ADN”.
Las muestras utilizadas para el proyecto Saca La Lengua se secuenciarán a través de la tecnología MiSeq de la empresa Illumina.
9 Secuenciación del ADN
Las técnicas de secuenciación de ADN permiten conocer las secuencias de nucleótidos (A-T-G-C) del ADN y su orden, es decir, permiten realizar una lectura del ADN.
La técnica de secuenciación utilizada en el proyecto Saca La Lengua es la técnica de Miseq desarrollada por la empresa Illumina. Se trata, de una tecnología de secuenciación del grupo de segunda generación, secuenciación de alto rendimiento o “next generation sequencing” (en inglés).
Así funciona la tecnología MiSeq de Illumina.
¿Quieres aprender más? ¡Hazlo tú mismo!
Para descubrir el mecanismo de secuenciación Sanger, incluido dentro de primera generación de secuenciación, puedes consultar esta información interactiva.
Conoce el funcionamiento de secuenciación Sanger.
Esta animación realiza una explicación acerca la tecnología que utiliza la empresa Illumina.
En este vídeo se explica la secuenciación por síntesis.
Y lo mismo sucede en el intervalo del minuto 2’05’’ al 2’51’’ del siguiente vídeo.
10 Procesamiento de datos – Introducción a la bioinformática
Durante un ciclo de trabajo del secuenciador MiSeq, que dura unas horas, se lleva a cabo una lectura de 25 millones de nucleótidos (o secuencia de letras A,T,G,C).
Aunque esta secuencia de nucleótidos procede del mismo fragmento de ADN, se realizan lecturas múltiples del mismo para obtener resultados más fiables estadísticamente.
Gracias a códigos especiales podemos distinguir 384 tipos de ADN diferentes, que se contrastarán con los fragmentos de ADN disponibles en las bases de datos.
Dado que ningún cerebro humano sería capaz de ordenar estos 25 millones de secuencias de letras utilizando tan solo papel y bolígrafo, los programadores utilizan programas especiales para poder realizar esta tarea. En esto, precisamente, consiste la bioinformática.
¿Quieres aprender más? ¡Hazlo tú mismo!
La identificación de especies y la relación entre ellas a través de un árbol filogenético constituye una parte esencial de la bioinformática, así como del proyecto Saca La Lengua.
Saca la Lengua utilizará esta herramienta para la identificación y clasificación de las bacterias que se encuentran en el microbioma de la boca.
¿Qué herramienta de bioinformática utilizamos? Prueba estos dos pasos con nosotros a través de esta página.
La revista europea “Science in School” propone este ejercicio. En el siguiente enlace se encuentran las soluciones.
Si estás interesado en familiarizarte con la rutina de trabajo de un bioinformático y las bases de datos, el Netherlands Bioinformatics Centre (NBIC) propone una serie de retos interesantes:
- Resolver un asesinato en inglés o en castellano descargando los pdfs siguientes
- Entender una enfermedad de la visión
Para llevar a cabo algunos de esos retos, son necesarios determinados programas específicos. En este enlace es posible descargarlos gratuitamente y consultar sus explicaciones.
El NBIC también ha desarrollado un guión para crear más retos bioinformáticos y clases de biología a través de la Guía Navigene.